
计算模拟平台
maxflowj9九游会老哥俱乐部交流的解决方案丨分子生成模型新发展:根据蛋白配体间相互作用构建分子生成模型
j9九游会老哥俱乐部交流的解决方案 | 2022-10-16 11:02
一、背景介绍
近年来,分子深度生成模型在药物从头设计中的应用引起了人们的广泛关注。数据驱动的分子深度生成模型通过学习大量分子结构数据来近似化学空间的高维分布。到目前为止,大多数的分子生成模型依赖于纯结构生成中的二维配体信息。在此,作者提出了一种新的分子深度生成模型,该模型采用循环神经网络,并结合配体-蛋白质相互作用指纹作为约束。该指纹图谱基于配体结合构象构建,代表了配体在蛋白质口袋中的三维结合模式。在目前的工作中,作者训练了带有交互指纹约束的生成模型,并与正常的rnn模型进行了比较。研究表明,在配体-蛋白质相互作用指纹图谱的约束下训练的模型有明显的趋势生成保持相似结合模式的化合物。该研究结果表明,相互作用指纹约束生成模型在靶向分子生成和类药物化学空间的指导探索方面具有潜在的应用价值。
二、实验方法
1、数据集:选择cdk2(细胞周期蛋白依赖激酶2)晶体结构(pdb id: 2r3m)和a2a(pdb id: 4ug2)作为蛋白质模型进行对接。对接研究采用schrödinger软件包(2020版)的glide模块。从chembl数据库中随机抽取30万个化合物作为训练集。对于每个组合,前10个对接姿势(基于glide评分)留作进一步分析。每个目标特定模型的训练集由300000个 chembl集和目标活性集组成。活性集是在chembl中记录的cdk2或a2a基因的活性化合物)。
2、相互作用指纹构建
配体-蛋白相互作用指纹图谱之前被提出用于表征配体的3d结合模式,并应用于虚拟筛选。在该研究中,作者开发了一个python脚本来解析配体对接姿势,从而生成配体ifp。作者构建了两种类型的ifp,即基于原子的ifp (aifp)和基于残基的ifp (resifp),分别在原子和残基水平上描述配体结合模式。如图1所示,首先识别组成结合口袋的残基或原子,然后检测蛋白质和配体原子之间的五种相互作用(即氢键、卤素键、静电、疏水和芳香相互作用)。每个蛋白质残基/原子有五种相互作用类型,即:五位。每个ifp位设置为“0”或“1”,表示特定残基/原子与配体之间没有或存在特定的相互作用类型。表1列出了定义这些交互类型的空间标准。如果每个化合物输出多个对接姿态,则不同对接姿态产生不同的ifp。
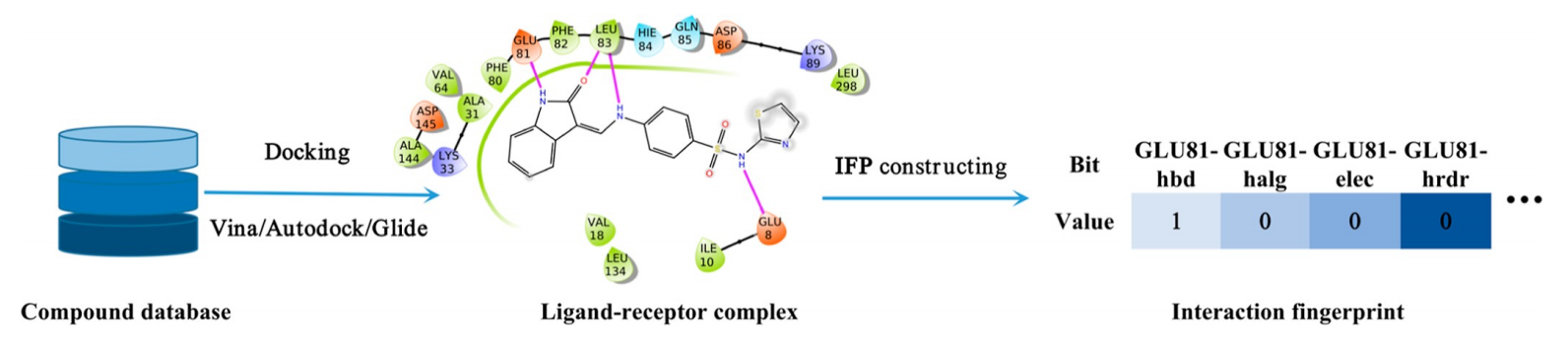
图1:交互指纹构建示意图及基于原子的交互指纹组成。交互类型包括氢键(hbd)、卤素键(halg)、芳香pi−pi相互作用(pipi)、静电相互作用(elec)、疏水相互作用(hrdr)。

表1:配体−蛋白相互作用检测参数
3、构建crnn模型
训练生成模型的基本工作流程如图2所示。首先,将化合物对接到特定的蛋白质结合位点,然后根据化合物对接姿态生成ifps作为约束条件,应用于crnn模型。利用kotsias等人开发的crnn模型在ifps约束下训练smiles, ifps表征配体3d结合模式。数据集按9:1分为训练集和验证集两部分,控制模块采用relu激活函数,由4个密集层组成。最后密集层的输出用来设置后面两层的初始隐态和细胞态lstm (长短期记忆)细胞。控制模块中每个密集层的大小为128,每个rnn层包含256个神经元。全部rnn层归一化。
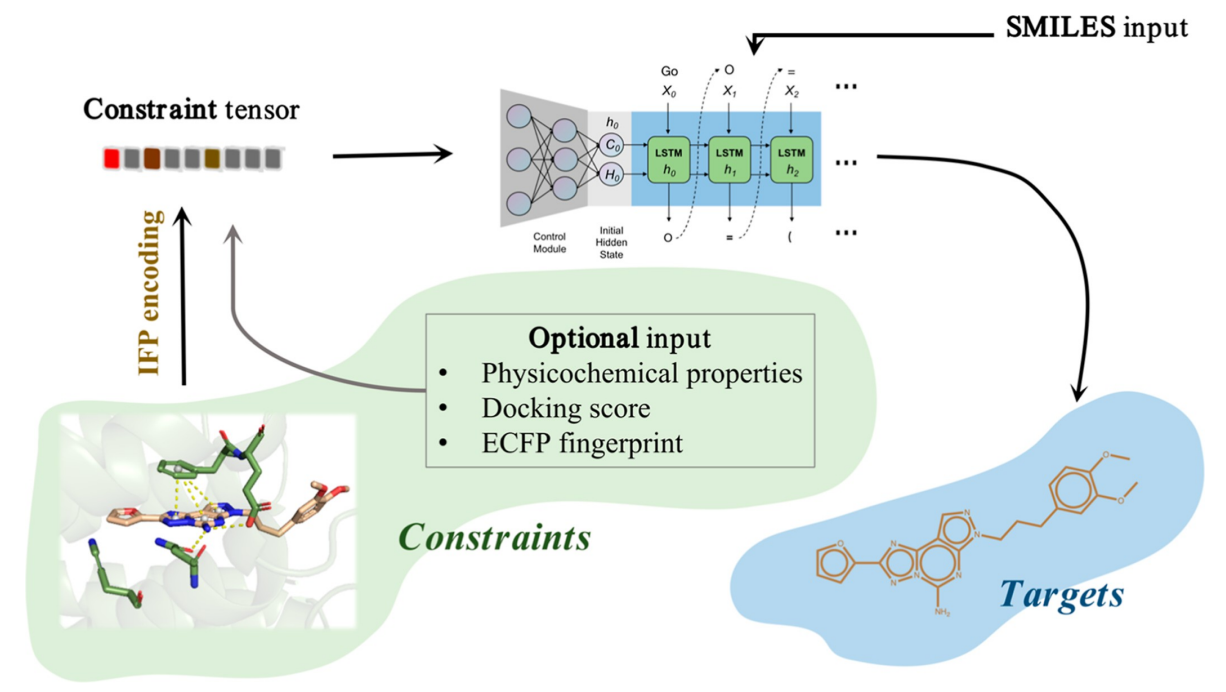
图2:基于ifp的crnn模型示意图。配体和受体之间的相互作用模式被编码到ifp载体中。然后,ifps被用作控制化合物生成的约束条件。
采用“监督学习”方法对crnn生成模型进行训练,以加快模型的收敛速度,提高模型稳定性。在“监督学习”方法中,在rnn序列生成的每一步中,都使用ground truth(地面真值)作为输入,而不是上一步预测的字符。使用rdkit包生成的256个规范的smiles字符串,在训练过程中使用默认参数设置的adam优化器,初始学习率为10−3。该模型通过自定义学习率计划训练了500个epoch。在前200个epoch,学习率保持不变,然后后面的300个epoch,在每个epoch后学习率呈指数递减,直到在最后的epoch时值为10 - 5。将预测字符与真实字符之间的交叉熵作为损失函数。
在分子生成阶段,对训练好的模型进行采样,在配体ifps的控制下生成smiles。将最后一层lstm的每个cell的输出向量设为一个表示smiles标记之间可能性分布的向量。在smiles生成过程中,使用多项采样从该向量中抽取每个单元的单个token,并在迭代过程中形成一个smiles字符串,直到对终止token进行采样。为了比较,作者还训练了一个基于rnn的在与基线生成模型相同的训练集上不运行强化学习的reinvent模型,该模型在正常的rnn模型上训练,不使用配体ifp约束生成结构(即不包括控制模块)。
4、模型构建的约束组合
本研究以配体-蛋白ifp作为主要的约束条件,在一定程度上直接反映了配体在结合位点上的结合方式。此外,其他信息,如对接分数,分子指纹(如ecfp指纹)和分子物理化学性质,包括logp,拓扑极性表面积(tpsa)、分子量(mw)、药物相似度(qed)、氢键受体数量(hba)和供体(hbd),结合ifp进行模型构建。此外,还探索了ecfp指纹作为指导结构生成的约束。在这里,ecfp使用了半径为3的1024比特摩根指纹。结合这些附加约束,试图更好地控制生成结构的质量,并检查crnn模型的鲁棒性。
三、实验结果
1、生成化合物的有效性
对样本256个化合物在训练过程中的有效性进行了检验,结果如图3所示。
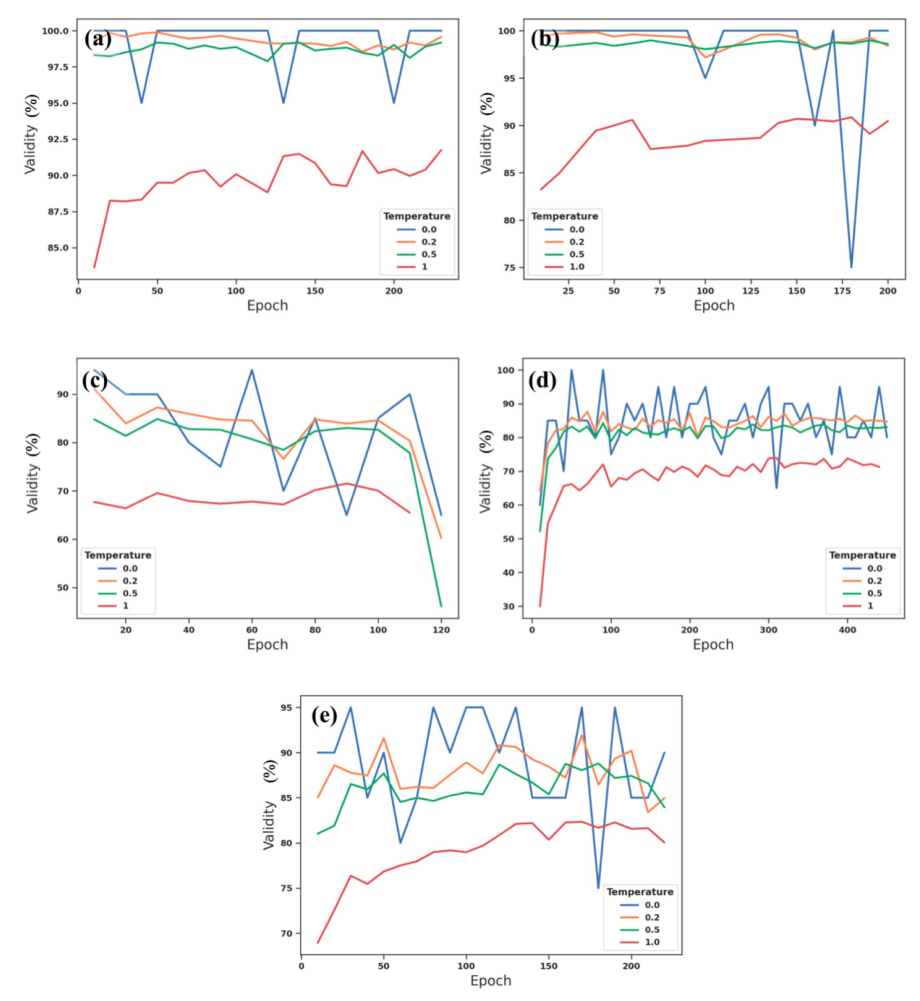
图3:生成的化合物在不同epoch和采样温度下的有效性。(a)aifp模型;(b) resifp模型;(c) dscorepp aifp模型;(d) dscorepp aifp模型(1个姿势);和(e) ecfp aifp模型。“0.2”、“0.5”和“1.0”分别为rnn模块。
2、cdk2的aifp模型
为了评价aifp模型的性能,作者选取了20个cdk2活性化合物的ifps作为种子采样特定的化学空间。由图4a可以看出,aifp化合物集合的对接得分略高于活性集合,且低于reinvent集合和随机集合,这表明aifp模型生成的化合物的对接得分高于reinvent集合和随机集合。这意味着约束模型可以在预测结合能方面产生更好的化合物。生成的化合物和种子化合物的分子相似性分布在图4b中,可以看到,这四组化合物的分布似乎都非常相似。如图4c,d所示,一般情况下,aifp模型生成的化合物ifp回收率 (rifp)和ifp谷本相似度(simifp) 最高。这表明aifp条件模型可以在所有集合中生成与种子原子ifp高度相似的化合物。
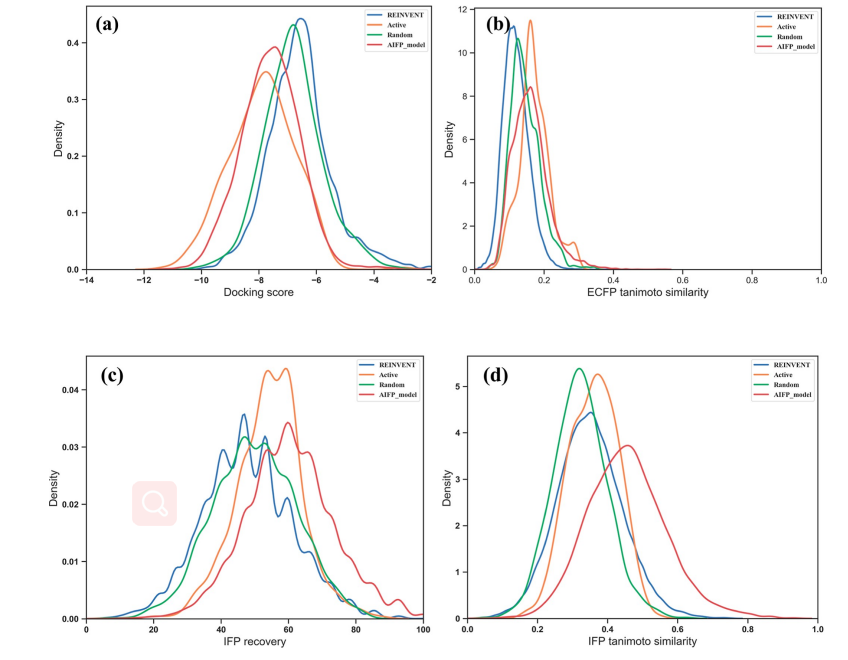
图4:评价指标在cdk2 aifp模型生成化合物中的分布。图例中的“reinvent”表示在chembl上训练的reinvent模型生成的化合物。“active”指chembl数据库中ic50值低于50 nm的cdk2活性化合物。“random”是指从chembl中随机选择的化合物集。
3、复合cdk2-constrained模型
对接得分在不同复合集之间的分布如图5a所示。与aifp模型相比,dscorepp aifp模型生成的化合物的对接得分进一步提高,与cdk2活性水平相同。这可能是由于在约束集中包含了对接分数。如图5b所示,active集和dscorepp aifp集与种子结构的相似性略高于reinvent集和随机集。如图5c,d所示,dscorepp aifp集达到最高所有化合物集合的aifp回收率和相似度。然而,加入复合约束后,aifp的富集效应在一定程度上被稀释,这可能是由于多个约束会引入冲突,导致ifp回收率和ifp谷本相似性降低。
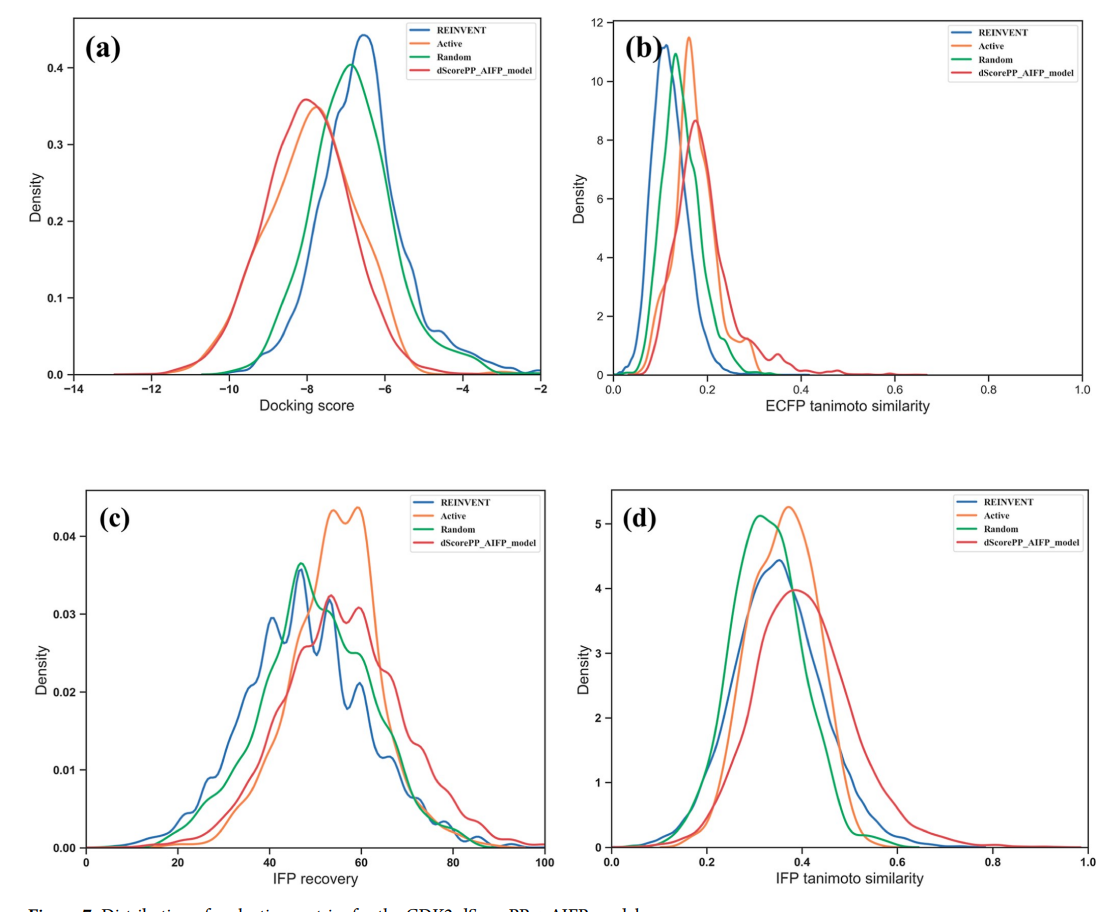
图5:cdk2 dscorepp aifp模型评估指标分布。
4、cdk2受体ecfp aifp模型
如图6b,c所示,由于加入了ecfp相似性约束,ecfp aifp模型与种子结构的ecfp相似性显著增加。同时,生成的ecfp aifp模型集仍然保持着最高的aifp位恢复率,以及最高的aifp与种子相似。与aifp模型的结果相比,ecfp aifp模型生成的集合似乎在一定程度上以失去多样性为代价,变得更接近它们的种子结构。
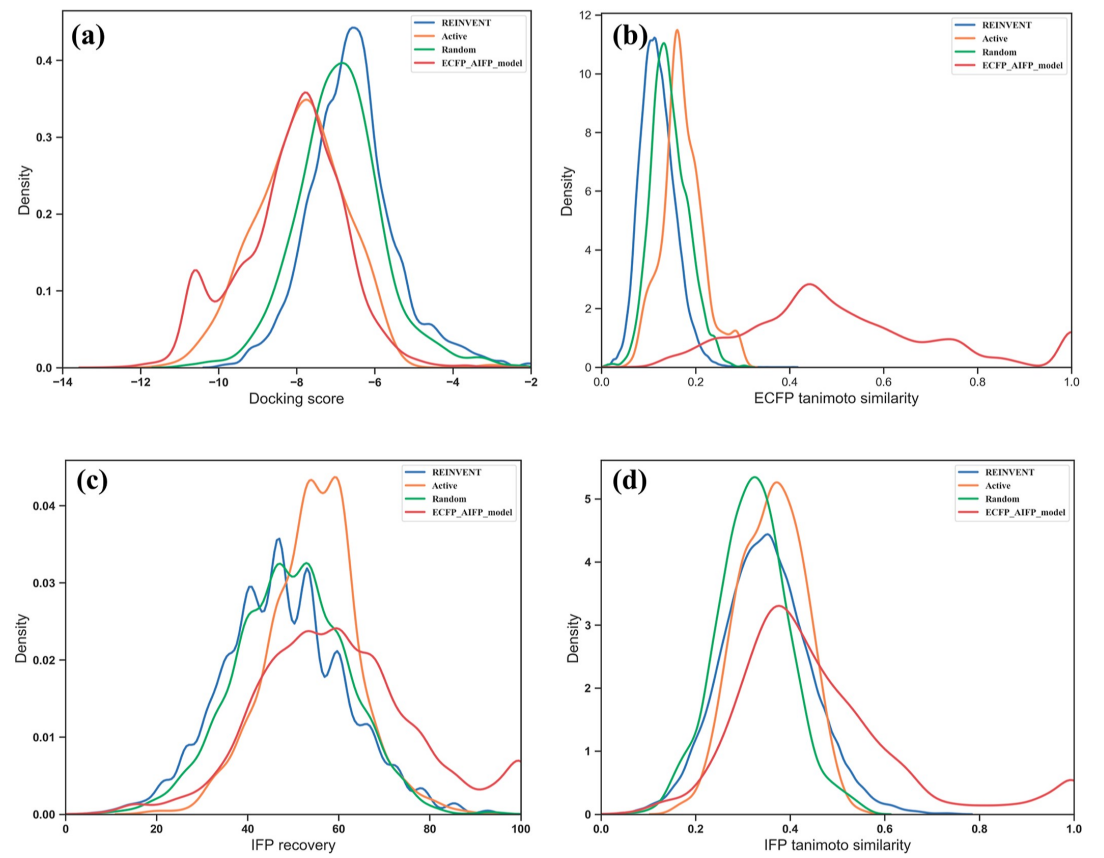
图6:cdk2 ecfp aifp模型生成化合物的评价指标分布。
5、模型平均性能的比较
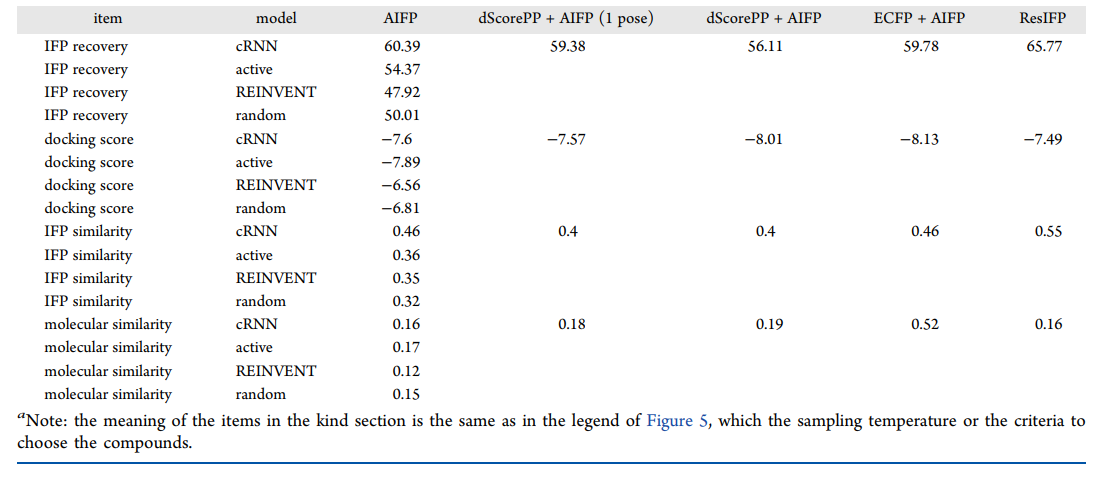
如表2所示,通过对接得分、ifp回收率、ifp相似度、与种子分子相似度等指标,比较了不同约束条件下分子深度生成模型的性能。在所有模型中,resifp和aifp模型的ifp回收率和相似度最高,而它们的分子相似度较低。另一方面,ecfp aifp模型在对接得分上排名第一,几乎在所有指标上都表现良好。
表2:基于所有生成化合物的cdk2深度生成模型的平均性能
四、实验结论
在本研究中,作者提出了一种利用配体结合模式信息的约束生成模型。配体/蛋白质相互作用指纹图谱用于表征配体结合模式,并作为侧信息纳入生成模型。探索其他分子理化性质以及对接得分,结合ifp作为建立模型的约束条件。从这些模型生成的化合物,平均而言,明显具有较高的比例具有种子化合物的理想结合模式,也往往具有较高的比例满足约束中定义的预定义标准。另一方面,一些生成化合物的骨架结构与种子化合物的骨架结构截然不同。该研究结果表明,这种嵌入生成模型的结合模式可能是一个有用的工具,以指导识别过程。该模型的一个限制是,模型的ifp输入高度依赖于分子对接位姿的质量,因此使用合适的对接算法来生成对接位姿是至关重要的。
参考文献:j. chem. inf. model. 2022, 62, 14, 3291–3306