

计算模拟平台
ds案例【酶设计】系列三:
基于分子模拟对mn-sod稳定性的研究
j9九游会老哥俱乐部交流的解决方案 | 2019-12-09 18:20
活性氧(reactive oxygen species, ros)是吞噬细胞发挥吞噬和杀伤作用的主要介质,它能迅速破坏包括dna、细胞膜和蛋白质在内的物质,造成组织损害。线粒体是活性氧的一个重要来源,会产生绝大多数超氧自由基,而锰超氧化物歧化酶(mn-sod)是唯一已知存在于线粒体内的抗氧化酶。由于sods的热稳定性和热变性会影响该酶发挥作用,因此迫切需要开发方法来提高该酶的稳定性。计算机辅助分子设计可以通过预测相应氨基酸在结构中的构象稳定性和折叠自由能来设计稳定的突变蛋白。
本文研究了通过突变提高mn-sod的热稳定性。文章借助discovery studio(ds)中的同源建模模块构建出mn-sod的三维结构,再通过虚拟氨基酸突变的方法设计了三类突变,包括单点突变,双点突变和三点突变。值得一提的是,研究者在选择突变氨基酸时,考虑了非保守的氨基酸,避免突变后导致酶自身失活。通过虚拟氨基酸突变结果,作者选择了三个突变体,分别为单点突变(sm-mnsod:glu215trp),双点突变(dm-mnsod:val140leu-glu215trp)和三点突变(tm-mnsod:val140leu-glu155trp-glu215trp)做后续研究。通过分子动力学模拟的方法可以对蛋白的稳定性做出更精准的判断,所以研究者利用ds的分子动力学模块进行了10ns的分子动力学计算,用以验证mn-sod野生型和突变株的稳定性。通过计算蛋白主链结构偏差(rmsd)可知野生型mnsod达到结构稳定状态时的最大平均距离偏差为1.98±0.17å,sm-mnsod为2.02±0.20å,dm-mnsod为1.61±0.17å,tm-mnsod为1.55±0.14å,说明双点突变和三点突变比野生型的结构更为稳定。最后研究者通过实验的方法对比了野生型和三个突变株在85℃孵化4个小时后的热稳定性,该结果和计算结果相吻合。说明通过计算机辅助设计的方法可以成功指导设计出高稳定性的mn-sod突变株。
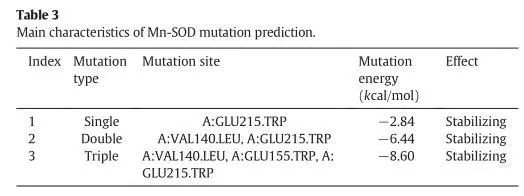
图一 虚拟氨基酸突变的计算结果
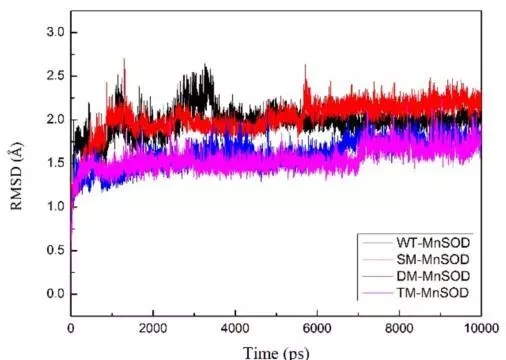
图二 分子动力学模拟的rmsd结果
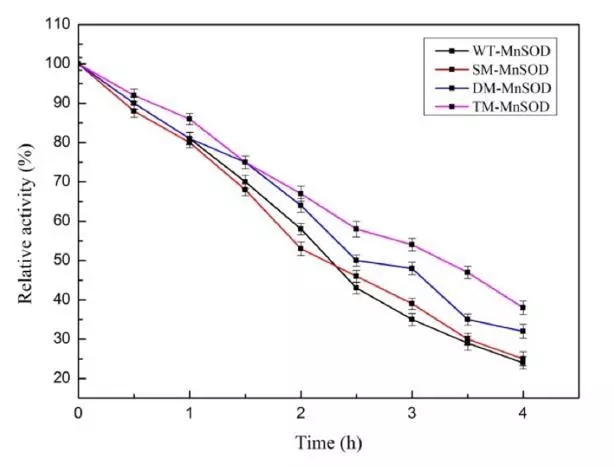
图三 野生型mn-sod及其突变株在85°c的热稳定性结果
分子动力学(molecular dynamics, md)是分子模拟中最常用的方法之一。该方法基于分子力场,能够动态的描述分子的运动状况,继而描述生命的动态过程。分子动力学在生命科学领域中的应用非常广泛,如蛋白质折叠的机理研究、酶催化反应的机理研究、与功能相关蛋白质的运动研究、生物大分子大范围构象变化的研究等等。近几十年来,分子动力学方法已经成功的运用于大分子体系低能量构象的模建、x射线晶体衍射以及nmr实验结果的处理。现在分子动力学方法已经成了理论生物学研究中必不可少的方法之一。
在discovery studio这一分子模拟的综合平台中,为用户提供了基于窗口的分子动力学模拟工具。使用ds,用户可以轻松的完成分子动力学的整个模拟过程(包括体系的准备、模拟过程的设置及结果的分析)。另外考虑到初级模拟者在整个模拟流程方面不熟悉,ds专门设置了一个“standard dynamics cascade”protocol。该protocol将整个动力学模拟的步骤集合到一个参数设置界面当中,方便初级模拟者的使用。