

资讯动态
neotrident news
资讯动态
由于受到新疾病出现的困扰,所以需要加快药物设计过程以开发针对这些疾病的新疗法。虽然基于深度学习的方法在基于配体的药物设计方面取得了一些成功。然而,这些方法在针对新靶点设计药物时面临数据稀缺的问题。在本项工作中,利用深度学习和分子模拟方法来开发药物设计流程,这对于目标特异性配体数据集有限或不可用的情况非常有用。在目标蛋白的活性位点筛选目标蛋白同源物的抑制剂,以获取初始目标特异性数据集。迁移学习用于学习特定目标数据集的特征。利用深度预测模型来预测新设计分子的潜在活性。使用强化学习将这两种模型结合起来,以设计具有优化对接分数的新化学实体。
ref:j. chem. inf. model. published: january 25, 2021 if=4.956
链接:
研究背景
新发疾病的爆发对人口构成严重威胁,并对国家的经济形势产生不利影响。尽管药物研发是治疗疾病最有希望的方法之一,但药物研发周期长、成本高与成功率低等三大风险。另外,传统的药物设计方法通过筛选公共或商业数据库中可用的化合物库或通过使用片段和药效团模型从头生成分子来识别特定于靶蛋白的分子。筛选过程不仅非常耗时,而且使用传统方法仅探索了化学空间的一部分。而深度学习方法可以弥合这一差距,并设计出具有所需类药物特性的多样化新化学实体。
随着人工智能和大数据领域的广泛应用,有可能从根本上改变药物发现中计算模型的准确性和可靠性。smiles表示或分子图表示通常用于训练深度神经网络模型以学习特征表示。药物发现还需要控制多个结构和物理化学参数。尽管早期的研究侧重于生成用于虚拟筛选的库,但引入用于特性优化的强化学习有助于使模型产生偏差,以生成目标特异性的化合物。此外,通过使用记忆增强神经网络,可以显着提高模型生成化学有效分子的效率。
在本项研究中,作者开发了一种从头药物设计的流程,可用于针对已知3d结构的新型靶蛋白。首先训练生成模型来学习已知类药物分子的语法,然后应用分子建模技术来编译特定于目标蛋白质的数据集,并将迁移学习用于学习编译数据集的特征。最后,强化学习被用来结合生成模型和预测模型,优化评分函数以产生可与靶标蛋白更好结合并具有所需药物特性的小分子。
药物设计流程
药物从头设计的流程图可分为以下几个步骤:
(1)数据集获取、预处理及训练分子生成模型:从chembl数据库下载分子,并以smiles格式表示,学习smiles语法的问题被转换为seq2seq问题。通过应用顺序过滤器对smiles数据集进行预处理,以除去立体化学、盐和具有不利原子或基团的分子,并删除重复项,仅收集长度≤100的分子。通过深度学习算法循环神经网络拟合出分子生成模型;
(2)迁移学习:将之前训练出的模型用小分子数据集进行再度训练,以通过迁移学习来捕获特定于目标受体的分子特征。而小分子数据集是以目标蛋白质的序列搜索相似蛋白质的小分子,并选择高活性分子,通过对接将这些分子对接在目标蛋白质的活性位点,进一步增强了该数据集的靶标选择性;
(3)训练预测模型:使用对接后打分的高和低对接分数的分子来训练预测模型,该模型则是学习小分子与其生物活性之间的关系;
(4)强化学习:使用强化学习将迁移学习后获得的生成模型与预测模型相结合,以生成目标特异性的化合物;
(5)分子过滤:生成的小分子通过理化性质过滤,并应用基于规则的过滤器除去具有不良基团的分子。具体流程图如图1

图1 基于深度学习药物从头设计的流程图
工作流验证
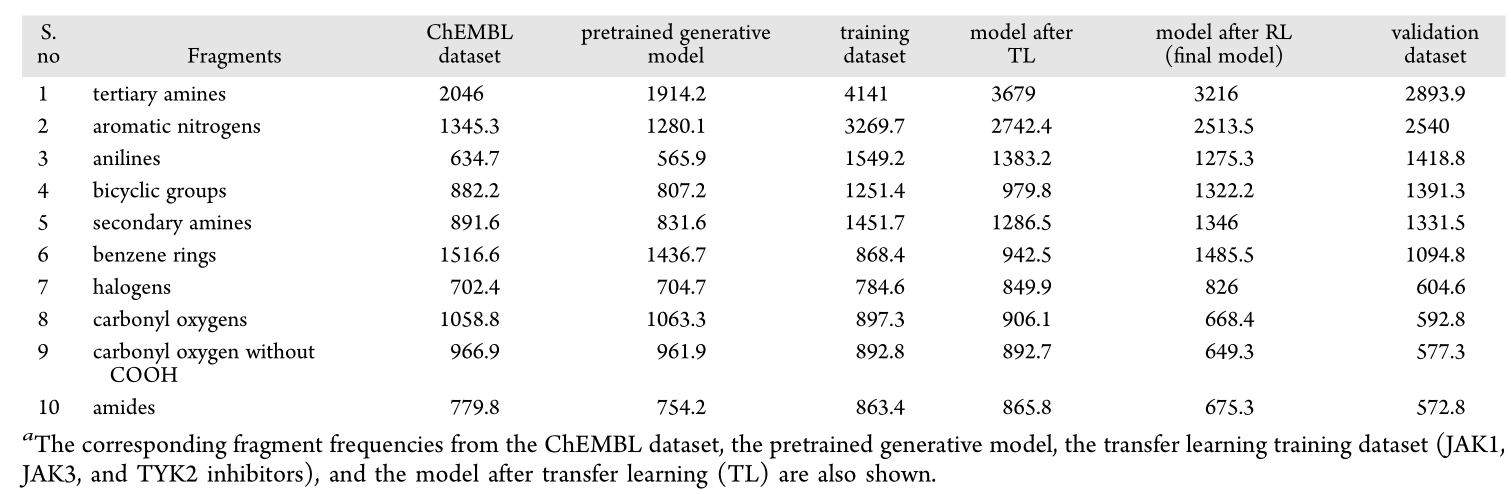
选择已知抑制jak1、jak3和tyk2蛋白的配体来构建靶标特异性训练数据集,设计高选择性jak2抑制剂。为了解所生成分子的亚结构特征,作者计算了每个batch的不同分子片段的频率,并计算所有batches的平均片段频率。如表1所示,所有前10个片段通常用于设计和合成高选择性jak2抑制剂。
表1 验证数据集中前10个分子片段的平均频率
为了进一步了解生成的分子相对于验证数据集的分布,图2a显示了较低维的embedding。尽管生成的分子与验证数据集的子集相似,但这些分子与jak2抑制剂相比具有更优的化学空间。这可以通过生成模型在增强学习后生成具有高于预期的对接得分和具有更好的理化性质的分子的能力来证实。将虚拟筛选后最终获得的6106个分子与特异jak2抑制剂的验证数据集进行比较可以发现,相对于来自验证数据集的分子,生成的分子集中的310个分子(5%)的tanimoto系数高于0.75。在这310个分子中,还发现一个分子与验证数据集相同(图2b)。基于虚拟筛选得分,与已知抑制剂分子相比,新分子可能是更好的jak2抑制剂,且这些代表性分子的子集如图2c所示。根据mm/pbsa计算,与现有的jak2抑制剂(-28.9 kj/mol)相比,新分子的预测结合自由能(mol_1285和mol_0717分别为-87.6和-75.3 kj/mol)表现更好。
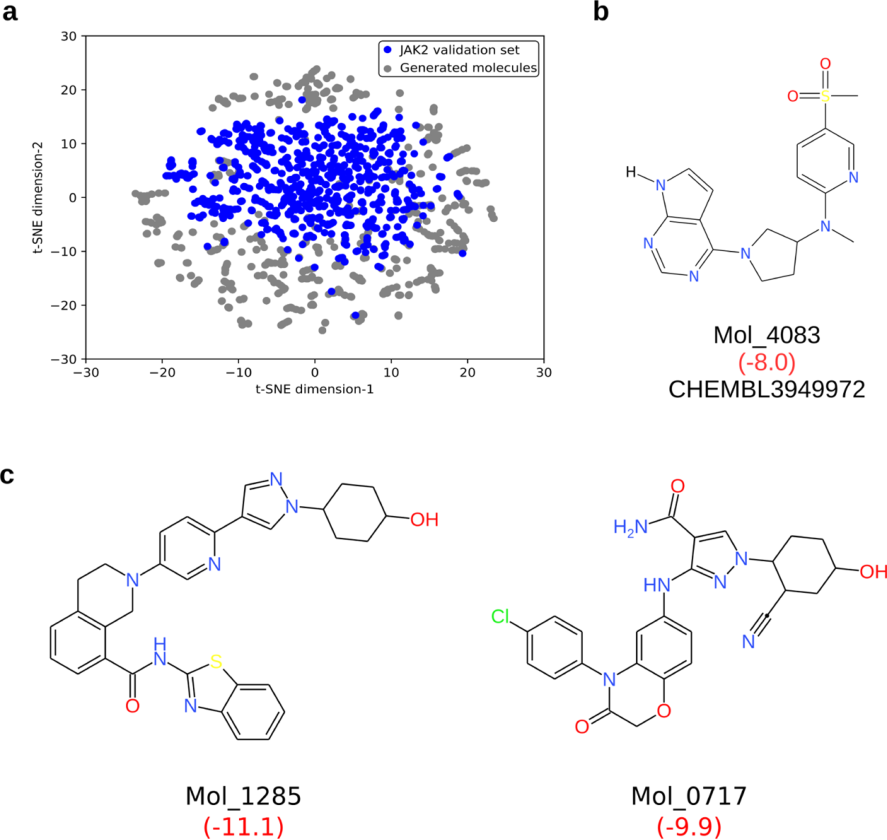
图2 生成与验证数据集相同的小分子和改进了对接分数的分子
总结
本文提出了一种从头药物设计方法,用于在没有靶标特异性小分子数据集的情况下生成针对该靶标的小分子。所提出的方法可用于结构且活性位点已知的靶蛋白,该蛋白的序列信息可被用于识别相关蛋白质,其抑制剂被用作目标特异性训练数据集。一旦满足这两个条件,所提出的方法可应用于任何靶蛋白。另外,此方法包含以下优势:
(1) 能够有效探索化学空间:传统的从头药物设计方法主要集中在与现有抑制剂具有高度骨架相似性的新型小分子的产生上,而深度学习模型显示了通过生成模型生成完全新颖的骨架和小分子的能力。从研究结果可以看到,与训练和验证数据集相比,所生成分子的骨架中有25.21%是新颖的;
(2) 靶标特定分子设计:通过使用迁移学习,深度学习模型能够从靶标特异性小分子数据集中捕获药效学表现形式;
(3) 物理化学性质的动态控制:可以使用增强学习将对接分数替换为所需的理化特性,以进行实时性能优化。
参考文献:
[1] krishnan s r, bung n, bulusu g, et al. accelerating de novo drug design against novel proteins using deep learning[j]. journal of chemical information and modeling, 2021, 61(2).
ref:j. chem. inf. model. published: january 25, 2021 if=4.956
链接:
研究背景
新发疾病的爆发对人口构成严重威胁,并对国家的经济形势产生不利影响。尽管药物研发是治疗疾病最有希望的方法之一,但药物研发周期长、成本高与成功率低等三大风险。另外,传统的药物设计方法通过筛选公共或商业数据库中可用的化合物库或通过使用片段和药效团模型从头生成分子来识别特定于靶蛋白的分子。筛选过程不仅非常耗时,而且使用传统方法仅探索了化学空间的一部分。而深度学习方法可以弥合这一差距,并设计出具有所需类药物特性的多样化新化学实体。
随着人工智能和大数据领域的广泛应用,有可能从根本上改变药物发现中计算模型的准确性和可靠性。smiles表示或分子图表示通常用于训练深度神经网络模型以学习特征表示。药物发现还需要控制多个结构和物理化学参数。尽管早期的研究侧重于生成用于虚拟筛选的库,但引入用于特性优化的强化学习有助于使模型产生偏差,以生成目标特异性的化合物。此外,通过使用记忆增强神经网络,可以显着提高模型生成化学有效分子的效率。
在本项研究中,作者开发了一种从头药物设计的流程,可用于针对已知3d结构的新型靶蛋白。首先训练生成模型来学习已知类药物分子的语法,然后应用分子建模技术来编译特定于目标蛋白质的数据集,并将迁移学习用于学习编译数据集的特征。最后,强化学习被用来结合生成模型和预测模型,优化评分函数以产生可与靶标蛋白更好结合并具有所需药物特性的小分子。
药物设计流程
药物从头设计的流程图可分为以下几个步骤:
(1)数据集获取、预处理及训练分子生成模型:从chembl数据库下载分子,并以smiles格式表示,学习smiles语法的问题被转换为seq2seq问题。通过应用顺序过滤器对smiles数据集进行预处理,以除去立体化学、盐和具有不利原子或基团的分子,并删除重复项,仅收集长度≤100的分子。通过深度学习算法循环神经网络拟合出分子生成模型;
(2)迁移学习:将之前训练出的模型用小分子数据集进行再度训练,以通过迁移学习来捕获特定于目标受体的分子特征。而小分子数据集是以目标蛋白质的序列搜索相似蛋白质的小分子,并选择高活性分子,通过对接将这些分子对接在目标蛋白质的活性位点,进一步增强了该数据集的靶标选择性;
(3)训练预测模型:使用对接后打分的高和低对接分数的分子来训练预测模型,该模型则是学习小分子与其生物活性之间的关系;
(4)强化学习:使用强化学习将迁移学习后获得的生成模型与预测模型相结合,以生成目标特异性的化合物;
(5)分子过滤:生成的小分子通过理化性质过滤,并应用基于规则的过滤器除去具有不良基团的分子。具体流程图如图1

图1 基于深度学习药物从头设计的流程图
工作流验证
选择已知抑制jak1、jak3和tyk2蛋白的配体来构建靶标特异性训练数据集,设计高选择性jak2抑制剂。为了解所生成分子的亚结构特征,作者计算了每个batch的不同分子片段的频率,并计算所有batches的平均片段频率。如表1所示,所有前10个片段通常用于设计和合成高选择性jak2抑制剂。
表1 验证数据集中前10个分子片段的平均频率

为了进一步了解生成的分子相对于验证数据集的分布,图2a显示了较低维的embedding。尽管生成的分子与验证数据集的子集相似,但这些分子与jak2抑制剂相比具有更优的化学空间。这可以通过生成模型在增强学习后生成具有高于预期的对接得分和具有更好的理化性质的分子的能力来证实。将虚拟筛选后最终获得的6106个分子与特异jak2抑制剂的验证数据集进行比较可以发现,相对于来自验证数据集的分子,生成的分子集中的310个分子(5%)的tanimoto系数高于0.75。在这310个分子中,还发现一个分子与验证数据集相同(图2b)。基于虚拟筛选得分,与已知抑制剂分子相比,新分子可能是更好的jak2抑制剂,且这些代表性分子的子集如图2c所示。根据mm/pbsa计算,与现有的jak2抑制剂(-28.9 kj/mol)相比,新分子的预测结合自由能(mol_1285和mol_0717分别为-87.6和-75.3 kj/mol)表现更好。

图2 生成与验证数据集相同的小分子和改进了对接分数的分子
总结
本文提出了一种从头药物设计方法,用于在没有靶标特异性小分子数据集的情况下生成针对该靶标的小分子。所提出的方法可用于结构且活性位点已知的靶蛋白,该蛋白的序列信息可被用于识别相关蛋白质,其抑制剂被用作目标特异性训练数据集。一旦满足这两个条件,所提出的方法可应用于任何靶蛋白。另外,此方法包含以下优势:
(1) 能够有效探索化学空间:传统的从头药物设计方法主要集中在与现有抑制剂具有高度骨架相似性的新型小分子的产生上,而深度学习模型显示了通过生成模型生成完全新颖的骨架和小分子的能力。从研究结果可以看到,与训练和验证数据集相比,所生成分子的骨架中有25.21%是新颖的;
(2) 靶标特定分子设计:通过使用迁移学习,深度学习模型能够从靶标特异性小分子数据集中捕获药效学表现形式;
(3) 物理化学性质的动态控制:可以使用增强学习将对接分数替换为所需的理化特性,以进行实时性能优化。
参考文献:
[1] krishnan s r, bung n, bulusu g, et al. accelerating de novo drug design against novel proteins using deep learning[j]. journal of chemical information and modeling, 2021, 61(2).