
计算模拟平台
q&a | 2020年7月discovery studio q&a精选
八月精选
j9九游会老哥俱乐部交流的解决方案 | 2020-07-24 15:14
每月初,创腾科技生命科学模拟技术部都会通过微信公众号定期总结 用户在软件使用过程中产生的一些具有代表性的问题。基于我们的工程师团队在分子模拟领域内十多年的经验,定期为大家带来实实在在的技术干货分享。
1. 如何从pdb数据库中选择蛋白晶体结构?
答:选择晶体结构的时候可以从以下几个方面加以考虑挑选:
a. 可以参考已发表的同体系模拟的文献中所选的晶体结构,文献年限可以是新一点的;
b. resolution值低的比较好;
c. 最好选带配体分子的复合物晶体结构,配体分子和对接的分子如果能比较类似则最为理想;
d. 结构是否完整,是否有缺失残基,缺失残基多还是少,最好选比较完整的结构。
2. 分子对接时如何定义活性位点?
答:分子对接确定结合位点的方法有三种。第一种是已知蛋白晶体,但晶体中无活性位点小分子,并且无相关实验和文献记录该蛋白的活性位点信息,这时可以选from receptor cavities这种方法,使用程序搜索蛋白的空腔进而对活性位点进行定义,但是这种方法定义的活性位点需要后期实验的验证;第二种是已知蛋白晶体,虽然晶体中无活性位点小分子,但是有相关实验和文献记录该蛋白的活性位点信息,这时可以选择from pdb site records这种方法,以关键氨基酸定义活性位点;第三种是下载的蛋白晶体中有配体可以选from current selection (先选中晶体中的小分子,再点击from current selection)。

3. 对接结果出现0 pose的原因以及如何解决?
答:对接结果出现0 pose时可以参考以下解决方法:
a. 体系是否正确预处理;
b. 定义的binding sphere位置是否正确,半径大小是否合适:该值只要保证有足够的空间供配体进行对接即可;
c. 使用高温动力学产生多构象时的步数:即random conformation下的dynamics steps参数项,增大该参数,可以使配体产生更合理的多构象;
d. 配体对接入蛋白活性位点之后的orientation参数:即maximum bad orientations和orientation vdw energy threshold两个参数,增大这两个参数(均改为999999),可以获得更多的对接pose。这两个参数的具体含义:orientation vdw energy threshold表示ligand在活性空腔内进行取向调整时范德华能的阈值,如果范德华能超过该值,那么这种orientation就不予以保留;maximum bad orientations表示在调整ligand取向时程序允许考察的取向的最大数目。
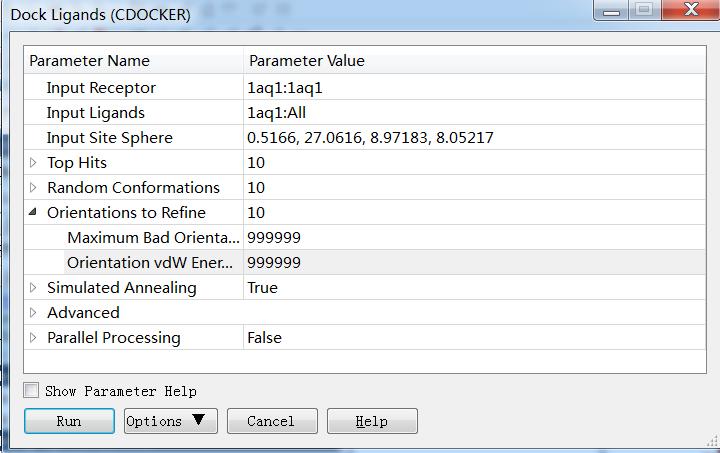
4. cdocker对接中,-cdocker_energy值为负值,而-cdocker_interaction_energy值为正值,应如何分析对接结果?
答:-cdocker_energy和-cdocker_interaction_energy理论上计算值应该都是正值,但是计算结果显示-cdocker_energy是负值,可能是因为分子内能过高,建议使用“minimize ligands”模块对小分子结构进行优化,释放一部分分子内能,然后再进行对接计算,以提高-cdocker_energy值。同时也可以观察蛋白结合口袋是否能容纳小分子配体,或采用protocols里的flexible docking加大蛋白对接时的柔性,用以找到合理的蛋白-小分子相互作用模式。另外cdocker_energy包含了配体分子内能和其与受体的相互作用能,cdocker_interaction_energy只包含相互作用能。如果不需要考虑分子间内能的因素,可以直接将-cdocker_interaction_energy做参考数据。
5. 分子对接中的alkyl、pi-alkyl、carbon hydrogen bond、pi-sulfur属于哪种类型的非键作用?
答:在discovery studio的help中可参考:help > help topics调取 > > non-bond interactions,有详细的描述可以参考,并且不同类型非键作用,所显示出的颜色也是不同的(如部分相互作用颜色的显示)。


6. 对接后如何把配体和蛋白一起导出作为一个复合物结构,然后进行动力学计算?
答:选中配体,按住鼠标左键,拖到pdb id上面后,放开左键,即可形成复合物。如先选中ibuprofen conformation 拖到1eqg上面。
