
计算模拟平台
ds案例 | 阿茨海默疾病模型中小神经胶质细胞调节剂的研究
biovia discovery studio 案例
j9九游会老哥俱乐部交流的解决方案 | 2021-01-27 11:36
鞘氨醇激酶1(sphk1)是一种依赖于乙酰辅酶a的乙酰基转移酶,在阿尔茨海默病(ad)模型中,其作用于神经元中的环氧合酶2(cox2)。但是,其背后作用机制目前尚不清楚。本研究结果表明n-乙酰鞘氨醇(n-as)首先由乙酰辅酶a和鞘氨醇通过sphk1生成。然后,n-as乙酰化cox2的丝氨酸565(s565),而n-as乙酰化的cox2诱导生成特异性促炎症消退介质(spm)。在ad的小鼠模型中,小神经胶质细胞显示n-as生成减少,从而导致乙酰s565 cox2和spm生成减少。用n-as处理可增加ad小鼠小神经胶质细胞中乙酰化的cox2和n-as触发的spm,导致神经炎症的消退,小神经胶质细胞吞噬作用的增加和记忆的改善。综上所述,这些结果确定了n-as在ad小神经胶质细胞功能障碍中的作用。
能量优化(minimization):中可基于charmm等一系列力场,以及包含gbsw等多种不同类型的隐式溶剂模型,对蛋白质、核酸、多糖、多肽、小分子以及相应的复合物进行能量优化。
ds cdocker 是基于charmm的对接程序,采用soft-core potentials以及optional grid representation将配体分子与受体活性位点进行对接。
ref:nature communications | (2020) 11:2358 if=12.121
链接:
环氧合酶2(cox2)是炎症反应的主要因素,催化其底物如花生四烯酸(aa),二十碳五烯酸(epa)和二十二碳六烯酸(dha)转化为促炎性脂质介质,包括前列腺素(pgs),11-羟基二十碳四烯酸(hete),11-羟基二十碳五烯酸或15-羟基二十碳五烯酸(hepe)和13-羟基二十二碳六烯酸(hdha)。有趣的是,当cox2被乙酰水杨酸(asa,阿司匹林)乙酰化时,乙酰化的cox2将其催化活性转换为将aa,epa和dha分别转化为15-hete,18-hepe和17-hdha。在存在5-脂氧合酶(5-lox)的情况下,这些分子可随后转化为15r-脂蛋白a4(15r-lxa4)和resolvins(rv)e1,d1,d2和d3,称为专门的前解析介体(spm)。spm具有强大的促分解作用,可导致免疫细胞浸润停止,促炎下调和消炎介质上调,并促进吞噬作用和组织再生。另外,最近的研究报告说,神经炎性疾病(例如阿尔茨海默氏病(ad))中的spm降低,从而导致小胶质细胞功能障碍。然而,在包括ad在内的大多数神经炎性疾病中,尚未确定通过spm调节小胶质细胞的基础机制。鞘氨醇激酶1(sphk1)是一种atp依赖性脂质激酶,可催化鞘氨醇转化为鞘氨醇-1-磷酸酯(s1p)。
最近,我们研究证明了sphk1是一种对cox2具有活性的乙酰辅酶a依赖性细胞质乙酰转移酶。研究还证实,ad大脑中神经元sphk1的水平降低,并且sphk1的增加通过乙酰化cox2的丝氨酸残基565(s565)促进了神经元(尤其是15r-lxa4)中spm的分泌,从而改善了ad样病理。但是,尚不完全了解sphk1介导的cox2乙酰化的生化和特异性ad致病机制。
本研究中为了进一步了解乙酰辅酶a与sphk1的作用机制,结合活性实验、突变实验和分子模拟方法使用2018中的cdocker将二者进行了分子对接,最终分析结果表明乙酰辅酶a结合在sphk1中的atp结合位点,二者是竞争性结合。乙酰辅酶a和sphk1中的鞘氨醇的反应产生n-乙酰鞘氨醇(n-as), sphk1产生的n-as与cox2结合,并使s565乙酰化,这对应于我们先前显示的被sphk1乙酰化的残基。 n-as乙酰化的cox2可提高15-hete,18-hepe和17-hdha的产量,这些产物可随后转化为spm,并提高了spm的产量,包括15r-lxa4,rve1和rvd1,类似于阿司匹林乙酰化cox2。此外,ad小胶质细胞中n-as的生成减少,从而导致乙酰s565 cox2和n-as触发的spm减少。
这些发现也使我们研究了n-as在ad小胶质细胞中的作用。通过n-as处理app / ps1小鼠,通过cox2的s565乙酰化作用,增加了小胶质细胞中n-as诱导的spm,从而直接调节了小胶质细胞的功能。最后,与未经处理的app / ps1小胶质细胞相比,n-as触发的spm导致神经炎症的缓解和与吞噬作用相关的几个反应性小胶质基因的上调,从而改善了ad病理。此外,淀粉样蛋白β(aβ)处理的人小胶质细胞也显示出n-as生成的减少,并且对这些人细胞进行n-as处理也提高了spm的产生和吞噬能力。总体而言,这些结果揭示了n-as的生物合成机制和功能,从而导致cox2的s565乙酰化和spm的产生。还揭示了n-as与小胶质细胞调节在ad发病机理中的关系,并提出了使用n-as或将来可能评估的相关衍生物治疗神经炎性疾病(如ad)的潜在疗法。
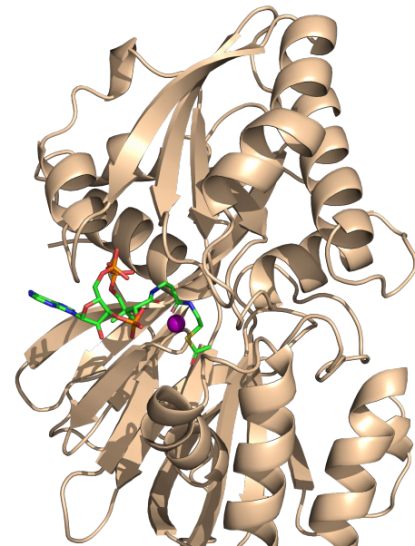
图一. acetyl-coa与sphk1蛋白的对接结果
为什么选择 biovia discovery studio?
1. ds cdocker 首先采用高温动力学的方法随机搜索小分子构象,随后采用模拟退火的方法将各个构象在受体活性位点区域进行优化,从而使对接结果更加准确;
2. biovia 中可基于charmm等一系列力场进行能量计算及能量优化;
3. biovia 应用广泛,操作简便,图形化界面十分友好,结果易于分析。